遗传算法解决TSP问题
来源:互联网 发布:电视定时开关机软件 编辑:程序博客网 时间:2024/05/17 01:31
原文:http://blog.csdn.net/mylovestart/article/details/8977005
遗传算法(Genetic Algorithm)是模拟达尔文生物进化论的自然选择和遗传学机理的生物进化过程的计算模型,是一种通过模拟自然进化过程搜索最优解的方法
遗传算法的基本运算过程如下:
a)初始化:设置进化代数计数器t=0,设置最大进化代数T,随机生成M个个体作为初始群体P(0)。
b)个体评价:计算群体P(t)中各个个体的适应度。
c)选择运算:将选择算子作用于群体。选择的目的是把优化的个体直接遗传到下一代或通过配对交叉产生新的个体再遗传到下一代。选择操作是建立在群体中个体的适应度评估基础上的。
d)交叉运算:将交叉算子作用于群体。所谓交叉是指把两个父代个体的部分结构加以替换重组而生成新个体的操作。遗传算法中起核心作用的就是交叉算子。
e)变异运算:将变异算子作用于群体。即是对群体中的个体串的某些基因座上的基因值作变动。
群体P(t)经过选择、交叉、变异运算之后得到下一代群体P(t 1)。
f)终止条件判断:若t=T,则以进化过程中所得到的具有最大适应度个体作为最优解输出,终止计算。
下面用C语言模拟遗传算法模拟TSP问题
TSP问题及旅行商问题,假设有一个旅行商人要拜访n个城市,他必须选择所要走的路径,路径的限制是每个城市只能拜访一次,而且最后要回到原来出发的城市。路径的选择目标是要求得的路径路程为所有路径之中的最小值
交叉的算法如下图
- #include<stdio.h>
- #include<string.h>
- #include<stdlib.h>
- #include<math.h>
- #include<time.h>
- #define cities 10 //城市的个数
- #define MAXX 100//迭代次数
- #define pc 0.8 //交配概率
- #define pm 0.05 //变异概率
- #define num 10//种群的大小
- int bestsolution;//最优染色体
- int distance[cities][cities];//城市之间的距离
- struct group //染色体的结构
- {
- int city[cities];//城市的顺序
- int adapt;//适应度
- double p;//在种群中的幸存概率
- }group[num],grouptemp[num];
- //随机产生cities个城市之间的相互距离
- void init()
- {
- int i,j;
- memset(distance,0,sizeof(distance));
- srand((unsigned)time(NULL));
- for(i=0;i<cities;i++)
- {
- for(j=i+1;j<cities;j++)
- {
- distance[i][j]=rand()%100;
- distance[j][i]=distance[i][j];
- }
- }
- //打印距离矩阵
- printf("城市的距离矩阵如下\n");
- for(i=0;i<cities;i++)
- {
- for(j=0;j<cities;j++)
- printf("%4d",distance[i][j]);
- printf("\n");
- }
- }
- //随机产生初试群
- void groupproduce()
- {
- int i,j,t,k,flag;
- for(i=0;i<num;i++) //初始化
- for(j=0;j<cities;j++)
- group[i].city[j]=-1;
- srand((unsigned)time(NULL));
- for(i=0;i<num;i++)
- {
- //产生10个不相同的数字
- for(j=0;j<cities;)
- {
- t=rand()%cities;
- flag=1;
- for(k=0;k<j;k++)
- {
- if(group[i].city[k]==t)
- {
- flag=0;
- break;
- }
- }
- if(flag)
- {
- group[i].city[j]=t;
- j++;
- }
- }
- }
- //打印种群基因
- printf("初始的种群\n");
- for(i=0;i<num;i++)
- {
- for(j=0;j<cities;j++)
- printf("%4d",group[i].city[j]);
- printf("\n");
- }
- }
- //评价函数,找出最优染色体
- void pingjia()
- {
- int i,j;
- int n1,n2;
- int sumdistance,biggestsum=0;
- double biggestp=0;
- for(i=0;i<num;i++)
- {
- sumdistance=0;
- for(j=1;j<cities;j++)
- {
- n1=group[i].city[j-1];
- n2=group[i].city[j];
- sumdistance+=distance[n1][n2];
- }
- group[i].adapt=sumdistance; //每条染色体的路径总和
- biggestsum+=sumdistance; //种群的总路径
- }
- //计算染色体的幸存能力,路劲越短生存概率越大
- for(i=0;i<num;i++)
- {
- group[i].p=1-(double)group[i].adapt/(double)biggestsum;
- biggestp+=group[i].p;
- }
- for(i=0;i<num;i++)
- group[i].p=group[i].p/biggestp; //在种群中的幸存概率,总和为1
- //求最佳路劲
- bestsolution=0;
- for(i=0;i<num;i++)
- if(group[i].p>group[bestsolution].p)
- bestsolution=i;
- //打印适应度
- for(i=0;i<num;i++)
- printf("染色体%d的路径之和与生存概率分别为%4d %.4f\n",i,group[i].adapt,group[i].p);
- printf("当前种群的最优染色体是%d号染色体\n",bestsolution);
- }
- //选择
- void xuanze()
- {
- int i,j,temp;
- double gradient[num];//梯度概率
- double xuanze[num];//选择染色体的随机概率
- int xuan[num];//选择了的染色体
- //初始化梯度概率
- for(i=0;i<num;i++)
- {
- gradient[i]=0.0;
- xuanze[i]=0.0;
- }
- gradient[0]=group[0].p;
- for(i=1;i<num;i++)
- gradient[i]=gradient[i-1]+group[i].p;
- srand((unsigned)time(NULL));
- //随机产生染色体的存活概率
- for(i=0;i<num;i++)
- {
- xuanze[i]=(rand()%100);
- xuanze[i]/=100;
- }
- //选择能生存的染色体
- for(i=0;i<num;i++)
- {
- for(j=0;j<num;j++)
- {
- if(xuanze[i]<gradient[j])
- {
- xuan[i]=j; //第i个位置存放第j个染色体
- break;
- }
- }
- }
- //拷贝种群
- for(i=0;i<num;i++)
- {
- grouptemp[i].adapt=group[i].adapt;
- grouptemp[i].p=group[i].p;
- for(j=0;j<cities;j++)
- grouptemp[i].city[j]=group[i].city[j];
- }
- //数据更新
- for(i=0;i<num;i++)
- {
- temp=xuan[i];
- group[i].adapt=grouptemp[temp].adapt;
- group[i].p=grouptemp[temp].p;
- for(j=0;j<cities;j++)
- group[i].city[j]=grouptemp[temp].city[j];
- }
- //用于测试
- /*
- printf("<------------------------------->\n");
- for(i=0;i<num;i++)
- {
- for(j=0;j<cities;j++)
- printf("%4d",group[i].city[j]);
- printf("\n");
- printf("染色体%d的路径之和与生存概率分别为%4d %.4f\n",i,group[i].adapt,group[i].p);
- }
- */
- }
- //交配,对每个染色体产生交配概率,满足交配率的染色体进行交配
- void jiaopei()
- {
- int i,j,k,kk;
- int t;//参与交配的染色体的个数
- int point1,point2,temp;//交配断点
- int pointnum;
- int temp1,temp2;
- int map1[cities],map2[cities];
- double jiaopeip[num];//染色体的交配概率
- int jiaopeiflag[num];//染色体的可交配情况
- for(i=0;i<num;i++)//初始化
- jiaopeiflag[i]=0;
- //随机产生交配概率
- srand((unsigned)time(NULL));
- for(i=0;i<num;i++)
- {
- jiaopeip[i]=(rand()%100);
- jiaopeip[i]/=100;
- }
- //确定可以交配的染色体
- t=0;
- for(i=0;i<num;i++)
- {
- if(jiaopeip[i]<pc)
- {
- jiaopeiflag[i]=1;
- t++;
- }
- }
- t=t/2*2;//t必须为偶数
- //产生t/2个0-9交配断点
- srand((unsigned)time(NULL));
- temp1=0;
- //temp1号染色体和temp2染色体交配
- for(i=0;i<t/2;i++)
- {
- point1=rand()%cities;
- point2=rand()%cities;
- for(j=temp1;j<num;j++)
- if(jiaopeiflag[j]==1)
- {
- temp1=j;
- break;
- }
- for(j=temp1+1;j<num;j++)
- if(jiaopeiflag[j]==1)
- {
- temp2=j;
- break;
- }
- //进行基因交配
- if(point1>point2) //保证point1<=point2
- {
- temp=point1;
- point1=point2;
- point2=temp;
- }
- memset(map1,-1,sizeof(map1));
- memset(map2,-1,sizeof(map2));
- //断点之间的基因产生映射
- for(k=point1;k<=point2;k++)
- {
- map1[group[temp1].city[k]]=group[temp2].city[k];
- map2[group[temp2].city[k]]=group[temp1].city[k];
- }
- //断点两边的基因互换
- for(k=0;k<point1;k++)
- {
- temp=group[temp1].city[k];
- group[temp1].city[k]=group[temp2].city[k];
- group[temp2].city[k]=temp;
- }
- for(k=point2+1;k<cities;k++)
- {
- temp=group[temp1].city[k];
- group[temp1].city[k]=group[temp2].city[k];
- group[temp2].city[k]=temp;
- }
- //处理产生的冲突基因
- for(k=0;k<point1;k++)
- {
- for(kk=point1;kk<=point2;kk++)
- if(group[temp1].city[k]==group[temp1].city[kk])
- {
- group[temp1].city[k]=map1[group[temp1].city[k]];
- break;
- }
- }
- for(k=point2+1;k<cities;k++)
- {
- for(kk=point1;kk<=point2;kk++)
- if(group[temp1].city[k]==group[temp1].city[kk])
- {
- group[temp1].city[k]=map1[group[temp1].city[k]];
- break;
- }
- }
- for(k=0;k<point1;k++)
- {
- for(kk=point1;kk<=point2;kk++)
- if(group[temp2].city[k]==group[temp2].city[kk])
- {
- group[temp2].city[k]=map2[group[temp2].city[k]];
- break;
- }
- }
- for(k=point2+1;k<cities;k++)
- {
- for(kk=point1;kk<=point2;kk++)
- if(group[temp2].city[k]==group[temp2].city[kk])
- {
- group[temp2].city[k]=map2[group[temp2].city[k]];
- break;
- }
- }
- temp1=temp2+1;
- }
- }
- //变异
- void bianyi()
- {
- int i,j;
- int t;
- int temp1,temp2,point;
- double bianyip[num]; //染色体的变异概率
- int bianyiflag[num];//染色体的变异情况
- for(i=0;i<num;i++)//初始化
- bianyiflag[i]=0;
- //随机产生变异概率
- srand((unsigned)time(NULL));
- for(i=0;i<num;i++)
- {
- bianyip[i]=(rand()%100);
- bianyip[i]/=100;
- }
- //确定可以变异的染色体
- t=0;
- for(i=0;i<num;i++)
- {
- if(bianyip[i]<pm)
- {
- bianyiflag[i]=1;
- t++;
- }
- }
- //变异操作,即交换染色体的两个节点
- srand((unsigned)time(NULL));
- for(i=0;i<num;i++)
- {
- if(bianyiflag[i]==1)
- {
- temp1=rand()%10;
- temp2=rand()%10;
- point=group[i].city[temp1];
- group[i].city[temp1]=group[i].city[temp2];
- group[i].city[temp2]=point;
- }
- }
- }
- int main()
- {
- int i,j,t;
- init();
- groupproduce();
- //初始种群评价
- pingjia();
- t=0;
- while(t++<MAXX)
- {
- xuanze();
- //jiaopei();
- bianyi();
- pingjia();
- }
- //最终种群的评价
- printf("\n输出最终的种群评价\n");
- for(i=0;i<num;i++)
- {
- for(j=0;j<cities;j++)
- {
- printf("%4d",group[i].city[j]);
- }
- printf(" adapt:%4d, p:%.4f\n",group[i].adapt,group[i].p);
- }
- printf("最优解为%d号染色体\n",bestsolution);
- return 0;
- }
3.1 编码
TSP问题编码一般有五种种不同的方式:
- 基于二进制的编码
- 基于矩阵的编码
- 基于邻接的编码
- 基于索引(Ordinary)的编码
- 基于路径的编码
基于二进制的编码是一种传统的编码方式,但是这种方式的编码,在经过遗传操作以后很难保证后代还是一个可行解,还需要另外的修正操作;基于矩阵的编码、邻接的编码以及索引的编码均比较复杂,并且需要占用大量的内存,应用的不是很广泛;目前最普遍用的编码方式就是基于路径的编码方式,因为它最直观最容易理解,操作起来也比较方便。以下将主要介绍基于路径的编码方式以及相关的遗传操作。
例如:
一个TSP问题,有六个城市{1,2,3,4,5,6},那么用基于路径的编码(1 3 5 4 6 2)就可以表示为一个个体。
3.2 交叉操作
下面主要介绍几种常见的交叉操作的方式。
3.2.1 部分匹配法(Partially Matching Crossover, PMX)
以两个父代个体为例:(1 2 3 4 5 6 7 8)和(2 4 6 8 7 5 3 1),随机选择两个交叉的点,假如第一个点为位置4,第二个交叉点为位置6,那么在两个点之间的位置将进行交叉,其它的位置进行复制或者用相匹配的数进行替换。在此实例中,第一个父代个体中4 5 6被选中,第二个父代个体中,8 7 5被选中。那么4 与8,5与7,6与5相匹配。匹配过程和如图2所示。
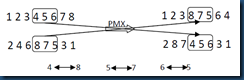
图2 PMX交叉操作
首先将4 5 6与8 7 5分别加入到子代2和子代1中相应的位置,然后将其他位置上的数字直接复制到相应的后代中,如果该数字已经在该子代中已经存在,则用相应的匹配法则进行替换,例如子代1中将7复制进去的时候,发现7已经存在在子代中,通过查找相应的匹配法则,发现,7与5匹配,然后复制5,又发现5也已经存在在该子代中,在此查找匹配法则,发现5与6匹配,将6复制,6不存在在该子代中,所以可以将6复制进去,如此反复,知道子代中城市的数目达到定义的长度,该子代创建完成。
3.2.2 循环交叉法(Cycle Crossover, CX)
依旧以两个父代个体为例:(1 2 3 4 5 6 7 8)和(2 4 6 8 7 5 3 1),在构造子代的过程中,首先从父代1中选取第一个元素,然后查找父代2中对应位置的元素在在父代1中的位置,将这个位置对应的元素假如到子代1中,如此过程反复,直到找到一个元素对应的父代2元素等于起始元素,次循环结束,然后将剩余未填充的位置用父代2相应的位置的元素进行填充,这样就可以完成子代1的创建。子代2创建方法类似。CX操作过程如图2所示。
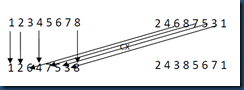
图3 CX操作
首先选择父代1中的第一个元素1,将它加入到子代1中,然后检查父代2中对应位置,该位置元素为2,在父代1中查找该元素,该元素在父代1中的位置为2, 将2加入到子代1的第二个位置,再次检查父代2中第二个位置的元素,它为4,然后查找它在父代1中的位置为4,将4加入到子代1的第四个位置,然后将其加入到子代1中对应的位置4,在检查父代2中该位置的元素,它为8,查找到它在父代1中的位置为8,然后将其加入到子代1中位置8,再次查找父代2中位置8的元素,它为1,等于起始选择的元素,循环结束,然后将子代1中3,5,6,7元素为空的位置,用父代2对应位置的元素进行填充,这些元素为6,7,5,3,所以得到的子代1为(1 2 5 4 7 5 3 8)。同样的方法,得到子代2为(2 4 3 8 5 6 7 1)。
3.2.3 次序交叉法1(Order Crossover, OX1)
还以两个相同的父代个体为例:(1 2 3 4 5 6 7 8)和(2 4 6 8 7 5 3 1),随机选择两个交叉的点,假如第一个点为位置3,第二个交叉点为位置5,那么在两个点之间的位置将进行交叉。然后从第二个交叉点开始,将原来相应的父代按照顺序进行填充,如果选择的元素已经存在在该子代中,跳过该元素,选择下一个元素,这种过程反复进行,知道所有的城市都被选择一次。在此实例中,第一个父代个体中3 4 5被选中,第二个父代个体中,6 8 7被选中。匹配过程和如图4所示。

图4 OX1操作
首先,将选择的位串进行替换,即3 4 5换到子代2中,6 8 7换到子代1中。现在从第二个交叉点开始,将父代1中的基因插入到子代1中, 所以,1 插入到第六个位置,2 插入到第七个位置,3插入到第八个位置,第二个交叉点后面已经填充满,将剩下的插入到第一个插入点前面的位置,所以4插入到第一个位置,5插入到第二个位置。这样,子代1构建完成。同样地方法,可以构建子代2.当遇到子代中已经存在的基因,就跳到下一个。
3.2.4 次序交叉法2(Order Crossover, OX2)
还以两个相同的父代个体为例:(1 2 3 4 5 6 7 8)和(2 4 6 8 7 5 3 1),随机选择几个交叉的点,假如第一个点为位置2,第二个交叉点为位置是3,第三个为位置6。首先找到父代2中相应位置的基因在父代1中位置,然后将其用父代2中选择的位置的基因进行替换插入到子代1中相应的位置中。然后将其它的位置用OX1相似的方法进行填充。这样就可以得到子代1.同样,替换角色,可以得到子代2。具体的操作过程如图5所示。
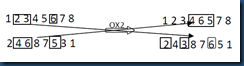
图5 OX2操作
首先找到父代2中第,2,3以及第六个位置的基因,它们为4,6和5,这三个基因在父代1中的位置为4,5和6,将它们替换成4,6,5,加入到子代1中相应的位置,然后将父代1中的基因按照顺序插入到子代1中,如果该基因已经存在在位串中,则跳过该基因,继续下一个。这样就可以构建完子代1。子代2也可以以相同的方法构造完成。
3.2.5 基于位置的交叉法(Position Based Crossover, POS)
还以两个相同的父代个体为例:(1 2 3 4 5 6 7 8)和(2 4 6 8 7 5 3 1),随机选择几个交叉的点,假如第一个点为位置2,第二个交叉点为位置是3,第三个为位置6。将两个父代中这些选择基因按照顺序交换,并且插入到子代1和2中。然后对其它位置的基因,按照顺序插入。具体操作过程如图6所示。

图6 POS操作
首先将2 3 6和4 6 8交换,分别插入到子代2和子代1相应的位置2,4,6中,然后将将父代1和父代2中的基因按照顺序插入到子代1和2中,如果该基因已经存在在位串中,则跳过该基因,继续下一个,知道位串的长度等于定义的长度。
3.2.6 交替位置交叉法(Alternating Position Crossover,APX)
以两个父代个体为例:(1 2 3 4 5 6 7 8)和(3 7 5 1 6 8 2 4),APX是一种简单的交叉操作方法,它是轮流选择父代1和2中的基因,直到子代的长度达到定义的长度为止。具体操作如图7所示。
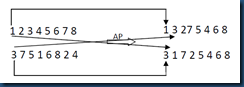
图7 APX操作
首先选择父代1中的第一个元素1,加入到子代1中,然后选择父代2中的第一个元素3,它不等于1所以也加入到子代1中,然后再选择父代1中的第二个元素2,它也不包含在当前的位串中,所以,也加入进去,然后再选择父代2中的第二个元素,……,直到子代1的长度达到8为止。同样的方法,可以得到子代2.
3.3 变异操作
同样地,下面介绍几种常见的变异操作方法。
3.3.1 替换变异(Displacement Mutation, DM)
DM先从父代个体中选择一个子位串,然后再随机在剩下的位串中选择一个为止,并插入该子位串,如图8所示。

图8 DM操作
3.3.2 交换变异(Exchange Mutation, EM)
EM是从父代个体中选择两个基因位置,然后互换这两个位置的基因。如图9所示。
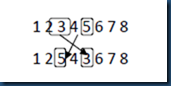
图9 EM操作
3.3.3 插入变异(Insertion Mutation, IM)
IM和DM类似,唯一的区别在于它是从父代个体中只选择一个基因,然后随机的插入到剩下的基因串中。如图10所示。

图10 IM操作
3.3.4 简单倒位变异(Simple Inversion Mutation, SIM)
SIM先从父代个体中选择一个基因串,然后将这个基因串中所有的基因倒位放置,如图11所示。
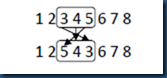
图11 SIM操作
3.3.5 倒位变异(Inversion Mutation, IVM)
IVM在SIM的基础上,随机选择一个插入位置,将这个到位后的基因串在插入到其中。如图12所示。
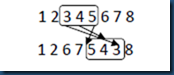
图12 IVM操作
3.3.6 争夺变异(Scramble Mutation, SM)
SM非常简单,先随机从父代个体中选择一个基因串,然后将除了第一个基因外的基因均向前移动一位,将第一个基因移到最后一位。具体操作过程如图13所示。

图13 SM操作
- 遗传算法解决TSP问题
- 遗传算法解决tsp问题
- 遗传算法解决TSP问题
- 遗传算法解决TSP问题
- 遗传算法解决TSP问题
- 遗传算法解决tsp问题
- 遗传算法解决TSP问题
- 遗传算法解决TSP问题
- 用遗传算法解决TSP问题
- 遗传算法解决网络布线问题 tsp
- 使用遗传算法解决TSP问题
- 利用遗传算法解决TSP问题
- 遗传算法解决TSP问题(C++)
- tsp问题——遗传算法解决
- 转:遗传算法解决TSP问题
- 遗传算法解决TSP问题笔记
- 初学遗传算法解决tsp问题(C++)
- 用遗传算法解决tsp问题
- HttpClient用POST上传文件
- linux 安装配置java环境
- Annotation自定义注解
- 文字匹配解析例子(读取Json 格式配置文件)
- TCP Incast学习之中遇到的问题
- 遗传算法解决TSP问题
- 聊天登陆之注册界面
- isodata算法确定k均值聚类的k值
- Objective-C中的instancetype和id区别
- 神秘的js执行顺序
- xshell配置
- NSIS脚本详解
- dom
- 用DIV做的页面在IE中不能居中,火狐中可以




