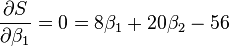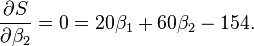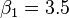R语言编程艺术——考试成绩的回归分析[一]
来源:互联网 发布:2.5平衡口耳机 知乎 编辑:程序博客网 时间:2024/05/18 02:30
1、R(至少)有三个各自独立的对象系统。S3和S4是S语言的不同版本,S3实现了基于generic function的面向对象。S4正式加入class definition等一套机制,使其更为严格。R5(reference class)是一种message passing OOP,更像Java。大部分基本统计方法和类(stats包)是用S3写的,Bioconductor是S4写的。
2、Bioconductor是一个基于R语言的、面向基因组信息分析的应用软件集合。Bioconductor的应用功能是以包的集成形式呈现在用户面前,它提供的软件包中包括各种基因组数据分析和注释工具,其中大多数工具是针对DNA微阵列或基因芯片数据的处理、分析、注释及可视化的。同时,Bioconductor还提供许多与DNA微阵列相关的数据包。
3、ExamsQuiz.txt,如下所示:
- 2.0 3.3 4.0
- 3.3 2.0 3.7
- 4.0 4.3 4.0
- 2.3 0.0 3.3
- 2.3 1.0 3.3
- 3.3 3.7 4.0
说明:
(1)数字表示的是学生成绩的学分绩点。每一行包含的是一个学生的数据,由其中考试成绩、期末考试成绩和平均小测验成绩组成。此例的兴趣点在于用期中考试成绩和平均小测验成绩来预测期末成绩。
(2)ExamsQuiz.txt数据集只是其中的部分。(自己没有找到完整的数据集)
(3)通过剖析整个案例,理解统计原理,提高R编程能力。
4、读入文件,如下所示:
- examsquiz <- read.table("ExamsQuiz.txt", header=FALSE)
(1)getwd()
解析:
查看当前工作目录。
(2)setwd(dir)
解析:
设置当前工作目录。dir是一个字符串,比如:"F:/R"。
(3)将ExamsQuiz.txt放在当前工作目录下面。
(4)这个数据文件的第一行不是记录的变量名,也就是说没有表头行,所以在函数调用中设定header=FALSE。实际上,表头参数的默认值已经是FALSE了,所以没有必要做前面那样的设定,不过这样做会更明了。语法如下:
- read.table(file, header = FALSE, sep = "", quote = "\"'",
- dec = ".", row.names, col.names,
- as.is = !stringsAsFactors,
- na.strings = "NA", colClasses = NA, nrows = -1,
- skip = 0, check.names = TRUE, fill = !blank.lines.skip,
- strip.white = FALSE, blank.lines.skip = TRUE,
- comment.char = "#",
- allowEscapes = FALSE, flush = FALSE,
- stringsAsFactors = default.stringsAsFactors(),
- fileEncoding = "", encoding = "unknown")
5、现在数据在examsquiz中,它是数据框类的R对象,如下所示:
- > class(examsquiz)
- [1] "data.frame"
6、为了检查数据文件刚才是否已读入,查看一下数据的前几行,如下所示:
- > head(examsquiz)
- V1 V2 V3
- 1 2.0 3.3 4.0
- 2 3.3 2.0 3.7
- 3 4.0 4.3 4.0
- 4 2.3 0.0 3.3
- 5 2.3 1.0 3.3
- 6 3.3 3.7 4.0
说明:
由于缺少数据表头行,R自动把列名设置为V1、V2和V3,行号出现在每行的最左边。
7、我们来用期中考试成绩(examsquiz的第一列)预测期末考试成绩(examsquiz的第二列),如下所示:
- lma <- lm(examsquiz[, 2] ~ examsquiz[, 1])
解析:
(1)这里调用lm()函数(lm是linear model的缩写),让R拟合下面的预测方程:
(2)我们用数据中的数对(期中考试成绩,期末考试成绩)拟合了一条直线。拟合过程是用经典的最小二乘法来完成的。
(3)也可以这样写:lma <- lm(examsquiz$V2 ~ examsquiz$V1)
8、lm()的返回结果现在是保存于变量lma中的对象。可以调用attributes()函数列出它的所有组件,如下所示:
- $names
- [1] "coefficients" "residuals" "effects"
- [4] "rank" "fitted.values" "assign"
- [7] "qr" "df.residual" "xlevels"
- [10] "call" "terms" "model"
- $class
- [1] "lm"
9、调用str(lma)可以得到lma的更详细说明。在命令提示符下键入系数的变量名就可以显示系数,如下所示:
- > lma$coefficients
- (Intercept) examsquiz[, 1]
- -1.293886 1.282751
解析:
在键入组件名时也可以使用缩写形式,比如:lma$coef。
10、打印对象lma,如下所示:
- > lma
- Call:
- lm(formula = examsquiz[, 2] ~ examsquiz[, 1])
- Coefficients:
- (Intercept) examsquiz[, 1]
- -1.294 1.283
解析:
为什么R只打印出这些项,而没有打印出lma的其他组件?这个问题的答案是,R在这里使用的print()函数是另一个泛型函数的例子,作为一个泛型函数,print()实际上把打印的任务交给了另一个函数——print.lm(),这个函数的功能是打印lm类的对象,即上面函数展示的内容。
11、可以用泛型函数summary()打印输出lma的更详细的内容,它实际上在后台调用了summary.lm(),得出针对某个特定回归模型的摘要,如下所示:
- > summary(lma)
- Call:
- lm(formula = examsquiz[, 2] ~ examsquiz[, 1])
- Residuals:
- 1 2 3 4 5 6
- 2.0284 -0.9392 0.4629 -1.6564 -0.6564 0.7608
- Coefficients:
- Estimate Std. Error t value Pr(>|t|)
- (Intercept) -1.2939 2.5308 -0.511 0.636
- examsquiz[, 1] 1.2828 0.8567 1.497 0.209
- Residual standard error: 1.497 on 4 degrees of freedom
- Multiple R-squared: 0.3592, Adjusted R-squared: 0.199
- F-statistic: 2.242 on 1 and 4 DF, p-value: 0.2087
12、lm()函数语法,如下所示:
- lm(formula, data, subset, weights, na.action,
- method = "qr", model = TRUE, x = FALSE, y = FALSE, qr = TRUE,
- singular.ok = TRUE, contrasts = NULL, offset, ...)
13、要用期中考试成绩和检测成绩预测期末考试成绩,可以使用记号+,如下所示:
- lmb <-lm(examsquiz[, 2] ~ examsquiz[, 1] + examsquiz[, 3])
解析:
+号并不表示计算两个量的和,它仅仅是预测变量(predictor variable)的分割符。
14、最小二乘法简介
解析:
最小二乘法(又称最小平方法)是一种数学优化技术。它通过最小化误差的平方和寻找数据的最佳函数匹配。利用最小二乘法可以简便地求得未知的数据,并使得这些求得的数据与实际数据之间误差的平方和为最小。
15、最小二乘法示例
解析:
某次实验得到了四个数据点 :
: 、
、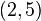 、
、 、
、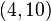 (上图中红色的点)。我们希望找出一条和这四个点最匹配的直线
(上图中红色的点)。我们希望找出一条和这四个点最匹配的直线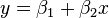 ,即找出在某种“最佳情况下”能够大致符合如下超定线性方程组的
,即找出在某种“最佳情况下”能够大致符合如下超定线性方程组的 和
和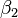 :
:
最小二乘法采用的手段是尽量使得等号两边的方差最小,也就是找出这个函数的最小值:
最小值可以通过对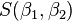 分别求
分别求 和
和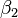 的偏导数,然后使它们等于零得到。
的偏导数,然后使它们等于零得到。
如此就得到了一个只有两个未知数的方程组,很容易就可以解出:
也就是说直线 是最佳的。
是最佳的。
参考文献:
[1]《R语言编程艺术》
[2]《R语言及Bioconductor在基因组分析中的应用》
[3] 最小二乘法:http://zh.wikipedia.org/zh-cn/%E6%9C%80%E5%B0%8F%E4%BA%8C%E4%B9%98%E6%B3%95
- R语言编程艺术——考试成绩的回归分析[一]
- R语言快速入门_案例分析之考试成绩的回归分析
- R语言系列—回归分析
- R语言编程艺术
- R语言-回归分析
- R语言回归分析
- R 语言与简单的回归分析
- R 语言与简单的回归分析
- R 语言编程艺术笔记
- 回归分析以及r语言实现(一)
- R语言_回归分析
- R语言-简单回归分析
- R语言中的回归分析
- R语言做回归分析
- R语言回归分析实例
- R语言与回归分析几个假设的检验
- R语言与回归分析几个假设的检验
- R语言回归分析中的异常值点的介绍
- HDU 1010 Tempter of the Bone
- Decorrelating Semantic Visual Attributes by Resisting the Urge to Share 论文笔记
- C++模板浅谈
- C++[算法]不借助第三个参数,交换两个数的值
- [Oracle] 分析函数(4)- Order By字句
- R语言编程艺术——考试成绩的回归分析[一]
- CDialog窗口类的Class Style中没有CS_VREDRAW和CS_HREDRAW导致自绘有问题
- 你妹都看得懂的手机网游制作教程(第21篇)游戏黑屏了,别急我们来调试代码
- C#,C++,Java三者的一些区别
- in和exists的区别与SQL执行效率分析
- 大数四则运算的C++实现
- 电视剧看合适的即可发生的方式根深蒂固
- ZBar 支持i386 armv6 armv7 armv7s x86_64 arm64
- TCP的那些事儿



![\begin{align}S(\beta_1, \beta_2) =& \left[6-(\beta_1+1\beta_2)\right]^2+\left[5-(\beta_1+2\beta_2) \right]^2 \\&+\left[7-(\beta_1 + 3\beta_2)\right]^2+\left[10-(\beta_1 + 4\beta_2)\right]^2.\end{align}](http://upload.wikimedia.org/math/2/d/9/2d9cfe8a47e91146c6e99b856fea042c.png)